前两天看到 Horticulture Research 发表的一篇文章:胡桃科基因组数据库。打开数据库链接使用他的blast功能会跳转到一个单独的页面,页面左上角SequenceServer字样,就去查了一下这个到底是啥?
SequenceServer 主页 http://sequenceserver.com/
我的理解这个工具的作用是在服务器端安装好这个软件后,可以通过浏览器使用blast工具比对服务器端构建好的数据库。比较神奇的是我在浏览SequenceServer主页时在最底部发现了两篇中文教程链接,点进去后竟然是简书里介绍SequenceSever使用的文章。这两篇文章的链接是:
https://www.jianshu.com/p/137ca16d6d5https://www.jianshu.com/p/de28be1a3bea
哈哈哈哈,不知道我写的这篇文章能不能被SequenceServer的主页收录呢?
下面开始介绍步骤首先安装miniconda
下载链接 https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh
下载好以后直接使用bash Miniconda3-latest-Linux-x86_64.sh一路默认就可以了
添加镜像
conda config –add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/pkgs/freeconda config –add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/conda-forgeconda config –add channels https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/biocondaconda config –set show_channel_urls yes
创建虚拟环境并安装需要的软件conda create -n punicaweb python=3.5conda activate punicawebconda install blastconda install ruby=2.4.2
ruby我最开始安装的版本是2.3.0,但是运行SequenceServer时遇到了报错
/home/miniconda3/envs/punicaweb/lib/ruby/2.3.0/rubygems/core_ext/kernel_require.rb:55:in `require’: /home/miniconda3/envs/punicaweb/lib/ruby/2.3.0/x86_64-linux/../../../libreadline.so.6: undefined symbol: PC – /home/miniconda3/envs/punicaweb/lib/ruby/2.3.0/x86_64-linux/readline.so (LoadError) from /home/miniconda3/envs/punicaweb/lib/ruby/2.3.0/rubygems/core_ext/kernel_require.rb:55:in `require’ from /home/miniconda3/envs/punicaweb/lib/ruby/gems/2.3.0/gems/sequenceserver-1.0.13/bin/sequenceserver:2:in `<top (required)>’ from /home/miniconda3/envs/punicaweb/bin/sequenceserver:22:in `load’ from /home/miniconda3/envs/punicaweb/bin/sequenceserver:22:in `<main>’
以上报错我也看不懂,我的解决办法是换ruby的版本碰运气,换到2.4.2后没有遇到报错
安装SequenceServergem install sequenceserver
这一步好像与网络有关系,有时候会遇到报错
Fetching: rack-1.6.13.gem ( 14%)ERROR: While executing gem … (Gem::RemoteFetcher::FetchError) too many connection resets (https://api.rubygems.org/gems/rack-1.6.13.gem)
如果遇到这个报错,就重新运行gem install sequenceserver我尝试了好多次才成功。######接下来是使用nginx设置反向代理
基本步骤
开放4567端口修改nginx的配置文件
具体操作可以参考我的这篇文章 https://www.jianshu.com/p/00c745728a64
构建blast需要的数据库makeblastdb -in output_1.fasta -dbtype prot -out PgCP -parse_seqids启动nginx/usr/local/nginx/sbin/nginx启动SequenceServer
我的blast数据库文件在/home/sequenceserver_practice/目录下
sequenceserver -d /home/sequenceserver_practice/
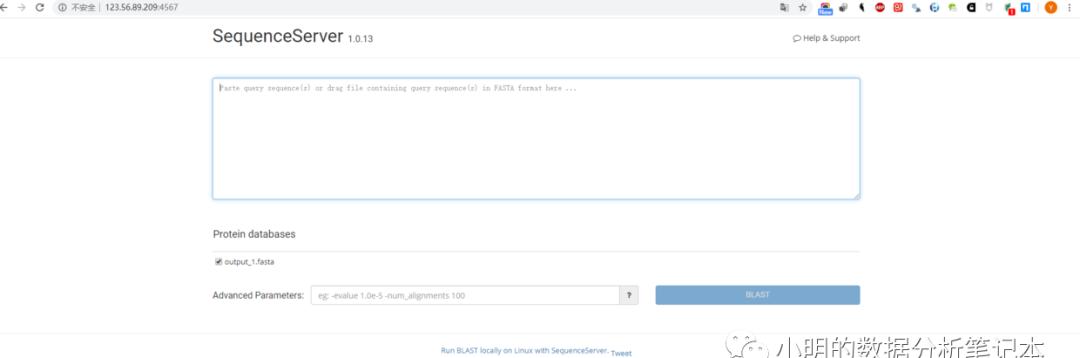
输入一条序列试一下,返回结果
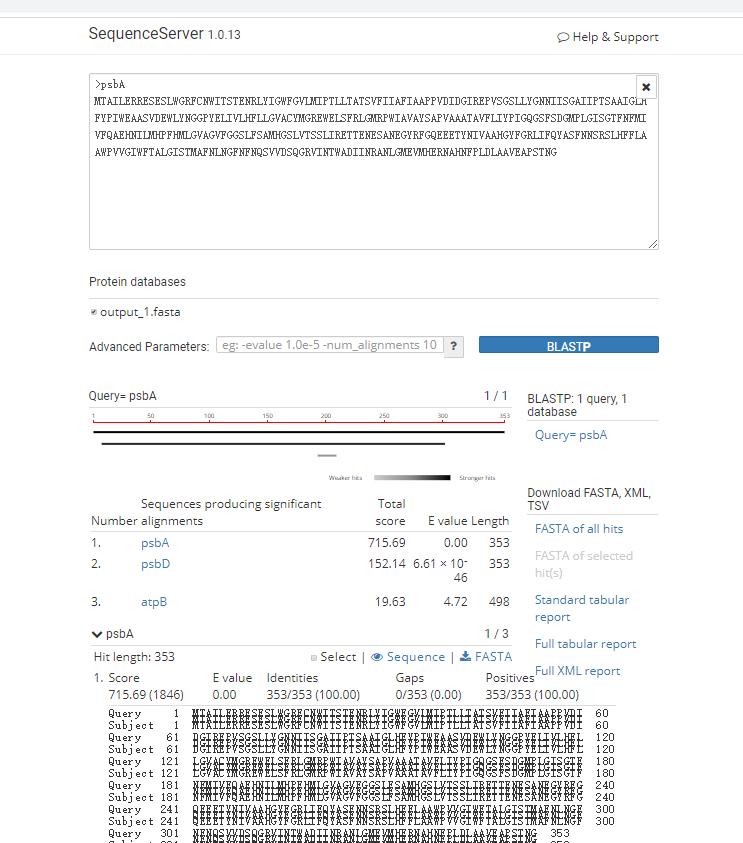
遇到一个问题是:如何限制输入序列的大小?
小明的数据分析笔记本